Amplían la información genética disponible de la langosta migratoria, causante de plagas devastadoras en África
Fuente: Universidad de Granada
Investigadores de la Universidad de Granada han ampliado la información genética disponible hasta la fecha de la langosta migratoria (Locusta migratoria), un ortóptero que causa plagas devastadoras en África por su gran movilidad, ya que es capaz de volar a gran velocidad con el viento (hasta 20 kilómetros por hora).
Los científicos, pertenecientes al departamento de Genética, han descubierto por primera vez 62 familias de ADN satélite en esta especie, al desarrollar un protocolo informático (satMiner) basado en los programas bioinformáticos RepeatExplorer y DeconSeq. Como prueba de la mejora que supone este protocolo para el ensamblaje de elementos repetitivos a partir de secuencias cortas de ADN generadas por las nuevas técnicas de secuenciación masiva, los autores también han encontrado 85 familias de ADN satélite en la planta Luzula elegans, que son más del doble de los descritos previamente mediante un análisis convencional por RepeatExplorer, y con abundancias más de 20 veces inferiores.
Los investigadores de la Universidad de Granada Francisco J. Ruiz-Ruano Campaña, María Dolores López León, Josefa Cabrero y Juan Pedro Martínez Camacho aplicaron satMiner a la búsqueda de satélites en la langosta migratoria, una especie donde, con anterioridad, no se había encontrado ni un solo ADN satélite.
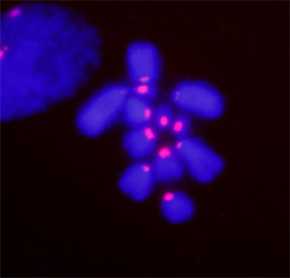
Imagen del satélite 1 (en rojo), que es el más abundante y se encuentra en todos los cromosomas de la langosta migratoria.
En un trabajo publicado en la revista Scientific Reports, los científicos de la UGR han propuesto, además, un nuevo término científico, “satelitoma”, para referirse al catálogo completo de ADNs satélites que existen en un mismo genoma, y han desarrollado métodos para poder estudiarlo en profundidad.
Hasta hace poco tiempo, se pensaba que los genomas eucariotas contenían sólo unos pocos tipos diferentes de ADN satélite, presentes en la heterocromatina de los cromosomas (es decir, en las regiones más condensadas). En pocos casos se habían descrito más de dos o tres satélites diferentes en una misma especie.
El mapeo físico de los ADNs satélites encontrados en el genoma de la langosta migratoria representa un hito importante porque nadie lo había hecho anteriormente para tantos satélites en una misma especie. Esto les ha permitido descubrir que su distribución sobre los cromosomas puede ser dispersa o estar clusterizada, es decir, muy concentrada en ciertas regiones cromosómicas, y que ello es independiente de la longitud del monómero que se repite. Además encontraron que más de la mitad de los satélites están clusterizados sobre un único cromosoma y, sorprendentemente, se encontraban presentes en decenas o cientos de contigs (segmentos de ADN superpuestos, que juntos representan una región consenso de ADN), diferentes del genoma de esta especie, sugiriendo que además se encuentran diseminados por el genoma.
Los autores de esta investigación esperan que el análisis en profundidad del satelitoma en otras especies, mediante estas nuevas metodologías, abra nuevas líneas de investigación sobre la evolución del ADN satélite a los niveles intra- e interespecífico, y que su utilización como marcadores de identificación cromosómica pueda ser de gran ayuda para completar las secuenciaciones genómicas en curso.
Finalmente, los científicos de la UGR esperan que el descubrimiento de tantos satélites nuevos contribuya a encontrar nuevas funcionalidades para esta parte mayoritariamente desconocida del repitoma.
Referencia bibliográfica:
High-throughput analysis of the satellitome illuminates satellite DNA evolution .Ruiz-Ruano, F.J., López-León, M.D., Cabrero, J. and Camacho, J.P.M. Scientific Reports 6, 28333, July 7, 2016, DOI:http://dx.doi.org/10.1038/srep28333
Contacto:
Juan Pedro Martínez Camacho
Catedrático del departamento de Genética de la UGR
Teléfonos: 958249708 – 958243262
Correo electrónico: jpmcamac@ugr.es
Últimas publicaciones
El bienestar animal así como un mayor rendimiento en cantidad y calidad han sido parte de los objetivos de este proyecto que ha contado como socios a COVAP, CETEMET, Keyter, Universidad de Córdoba y Cooperativas Agro-alimentarias de Andalucía.
Sigue leyendoEl grupo operativo INNOFINO, formado por el Consorcio ceiA3 junto a las universidades de Cádiz y de Córdoba, varios consejos reguladores andaluces así como distintas bodegas del marco de Jerez, acaban de presentar los resultados finales del proyecto en la Consejería de Agricultura, Pesca y Desarrollo Rural de la Junta de Andalucía en Sevilla.
Sigue leyendoInvestigadores de la Universidad de Sevilla y la empresa Civiencia, en colaboración con medio millar de participantes de distintos colectivos sociales, han desarrollado una plataforma digital con el objetivo de actuar contra las noticias falsas. Esta iniciativa está apoyada por la Oficina de Ciencia Ciudadana de Andalucía, que coordina la Fundación Descubre-Consejería de Universidad, Investigación e Innovación y la Universidad Pablo de Olavide y pretende potenciar la utilización de esta metodología entre distintos agentes de la región.
Sigue leyendo




