Andalucía ha secuenciado ya 25.000 muestras de coronavirus para el control de la enfermedad
La secuenciación genómica realizada por científicos andaluces permite establecer un mapa epidemiológico y realizar un estudio filogenético del virus, mostrando las mutaciones que experimenta a lo largo del tiempo, sus características y sus posibles relaciones con variantes de otros países o localidades, lo que facilita detectar las introducciones de virus y las cadenas de transmisión.
Fuente: Junta de Andalucía
Científicos andaluces han secuenciado, tras más de dos años de pandemia, 25.000 genomas de coronavirus para el control de la enfermedad, su transmisión y las distintas variantes, lo que permite mantener una estrecha vigilancia epidemiológica sobre el virus y la enfermedad que causa.
El circuito estable y bien estructurado puesto en marcha al inicio de la pandemia por la Dirección General de Salud Pública y Ordenación Farmacéutica, con el soporte del Servicio Andaluz de Salud, ha permitido identificar y analizar las muestras, tomadas directamente de pacientes infectados por el coronavirus causante del Covid-19. Actualmente la variante Ómicron es prácticamente dominante en Andalucía, con tres linajes mayoritarios BA.2, BA.2.9 y BA.1.1. Pero el sistema de vigilancia, desde su puesta en marcha hace dos años, ha registrado la presencia de las variantes más comunes (alfa, beta, delta, ómicron, etc.) y de 193 de sus linajes y sublinajes a lo largo de toda la pandemia, conteniendo así un registro epidemiológico extremadamente detallado que da cuenta del paso del virus por Andalucía.
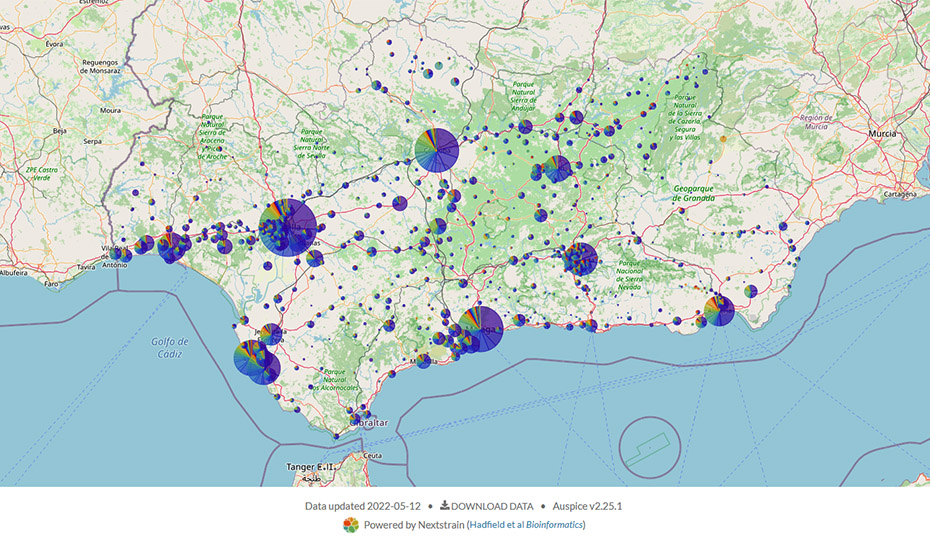
El circuito, que se expone en los próximos días en el marco del XXV Congreso de la Sociedad Española de Enfermedades Infecciosas y Microbiología Clínica, se enmarca en la integración de la secuenciación genómica del SARS-CoV-2 en las tareas de vigilancia realizadas por el Sistema de Vigilancia Epidemiológica de Andalucía. Se trata de una red colaborativa estructurada para conocer cómo se ha transmitido el coronavirus en Andalucía desde los inicios, cuáles han sido las zonas con un mayor índice de contagio y también cómo han influido las medidas sanitarias que se han ido implantado, entre otros aspectos.
Además, gracias a la secuenciación genómica, se puede realizar un estudio filogenético del virus, mostrando las mutaciones que experimenta a lo largo del tiempo, sus características y sus posibles relaciones con variantes de otros países o localidades, lo que facilita detectar las introducciones de virus y las cadenas de transmisión. Los resultados de la progresión de este estudio están disponibles públicamente en la página web: https://www.clinbioinfosspa.es/COVID_circuit/.
La secuenciación de los genomas de los virus está centralizada en los dos hospitales de referencia designados para ello: el Hospital Virgen del Rocío de Sevilla, donde se secuencian las muestras que proceden de centros hospitalarios y de Atención Primaria de la zona occidental de Andalucía (las provincias de Cádiz, Córdoba, Huelva y Sevilla); y el Hospital San Cecilio de Granada, para las que proceden de la zona oriental (Almería, Granada, Jaén y Málaga). La información obtenida se analiza en el Área de Bioinformática Clínica de la Fundación Progreso y Salud, en donde se interpreta y se configura un mapa epidemiológico del virus. Estos trabajos han sido impulsados por la Secretaría General de I+D+i en Salud.
Secuenciar el genoma del virus «es como leer el libro de instrucciones para descifrar su funcionamiento», explica Joaquín Dopazo, director del Área de Bioinformática Clínica. «Esto permite comparar cada libro de instrucciones y ver semejanzas y diferencias entre ellos que se pueden relacionar con su mayor o menor transmisibilidad o incluso con diferentes sintomatologías de los pacientes», señala.
Un circuito estable y coordinado
Hasta llegar a los dos centros de referencia, tanto los profesionales de Microbiología como los clínicos de los hospitales y de los centros de Atención Primaria son eslabones de la cadena que trabajan de forma coordinada con el Sistema de Vigilancia Epidemiológica de Andalucía, integrado por los profesionales de Salud Pública de los distritos y hospitales, Epidemiología de las Delegaciones Territoriales Provinciales y el Servicio de Vigilancia y Salud Laboral de la Dirección General de Salud Pública y Ordenación Farmacéutica.
Al final de esta cadena, el último eslabón es el Área de Bioinformática Clínica de la Fundación Progreso y Salud, donde un equipo de profesionales, entre los que se encuentran Javier Pérez Florido, Carlos Sánchez Francisco Ortuño, Jose Luis Fernández, y María Lara, analizan los datos obtenidos de la secuenciación, reportan a los centros de referencia los resultados para que estos informen al Ministerio de Sanidad de los hallazgos en la comunidad andaluza y los incorporan a las herramientas bioinformáticas para su interpretación más detallada.
Últimas publicaciones
Un equipo de investigación de la Universidad de Cádiz ha desarrollado un modelo matemático para estimar con mayor exactitud la cantidad de sedimento necesario para regenerar las costas. La propuesta permite ajustar mejor el volumen añadido al litoral, reduciendo costes y respetando la composición natural del ecosistema.
Sigue leyendoLa participación está abierta a: municipios de menos de 50.000 habitantes, así como a colectivos sociales y en riesgo de exclusión.
Sigue leyendoUn equipo de la Universidad de Sevilla descubre una diversidad genética inesperada en un grupo de tomillos ibéricos. El trabajo, basado en técnicas genómicas de última generación, revela linajes ocultos y plantea nuevos retos para la conservación de estas plantas tan emblemáticas.