Analizan miles de genomas de la iraquibacteria, causante de infecciones oportunistas en ambientes hospitalarios
Según este estudio de la Universidad Pablo de Olavide, el control de los sistemas de inmunidad bacterianos podría suponer un arma muy poderosa en la lucha futura contra las infecciones de A. baumannii.
Fuente: Universidad Pablo de Olavide
El grupo UPOBioinfo, liderado por el profesor de la Universidad Pablo de Olavide Antonio J. Pérez-Pulido, junto al grupo de Enfermedades Infeccionas del Instituto de Biomedicina de Sevilla y el Hospital Universitario Virgen del Rocío, y a un equipo de investigadores del Data Science & Big Data Research Lab de la UPO, entre los que se incluyen Federico Divina y Miguel García, emprendieron en 2018 el análisis de casi 2.500 genomas de A. baumannii. El proyecto, enmarcado en la tesis de Eugenio L. Mangas, ha publicado los primeros resultados en la revista especializada de la Sociedad Europea de Microbiología, Microbial Genomics, definiendo por primera vez el conjunto completo de genes que presenta la bacteria y distinguiendo dos tipos diferentes de cepas.

De izquierda a derecha: Jerónimo Pachón, Mª Eugenia Pachón, Eugenio L. Mangas, Antonio J. Pérez-Pulido, Alejandro Rubio y Fererico Divina.
“Por un lado, existen cepas que contienen un menor número de genes, lo cual podría deberse a los sistemas de inmunidad que presentan. En particular, estas cepas contienen sistemas llamados CRISPR/Cas, los cuales se conocen desde hace relativamente poco tiempo y precisamente permitirían a la bacteria ‘defenderse’ de la entrada de nuevos genes. El otro grupo de cepas no tiene apenas sistemas de inmunidad y eso parece que les ha permitido tener un mayor número de genes, sobre todo provenientes de unas estructuras similares a minicromosomas, denominadas plásmidos”, explica Antonio J. Pérez Pulido. Estas cepas, hipotéticamente podrían adquirir más fácilmente genes de virulencia y resistencia a antibióticos, por lo que, como subraya el investigador, el control de estos sistemas de inmunidad bacterianos podría suponer un arma muy poderosa en la lucha futura contra las infecciones de A.baumannii.
Durante el proceso de trabajo, para el análisis de los casi 2.500 genomas, según detalla Antonio J. Pérez Pulido, fue necesario el uso del supercomputador del Centro de Cálculo Científico de la UPO (C3UPO), cuyo administrador actual, Alejandro Rubio, puso en marcha diversos experimentos in silico.
A. baumannii, entre las bacterias para las que urge el desarrollo de medicamentos
La Organización Mundial de la Salud publicó en 2017 la lista de bacterias para las que urgía el desarrollo de nuevos medicamentos y dentro de ella, Acinetobacter baumannii fue situada en el grupo de prioridad crítica, junto con Pseudomonas aeruginosa y la familia Enterobacteriaceae. En concreto, A. baumannii es una bacteria causante de infecciones oportunistas en ambientes hospitalarios. De hecho, esta bacteria es responsable de más del 10% de las infecciones intrahospitalarias, y causa diversas enfermedades como neumonía, bacteriemia, infecciones de la piel y tejidos blandos, endocarditis, infecciones del tracto urinario y meningitis secundaria, entre otras. Afecta sobre todo a individuos inmunodeprimidos u hospitalizados en las unidades de cuidados intensivos (UCI) y se estima que cada año produce un millón de infecciones en todo el mundo, causando alrededor de 15.000 muertes.
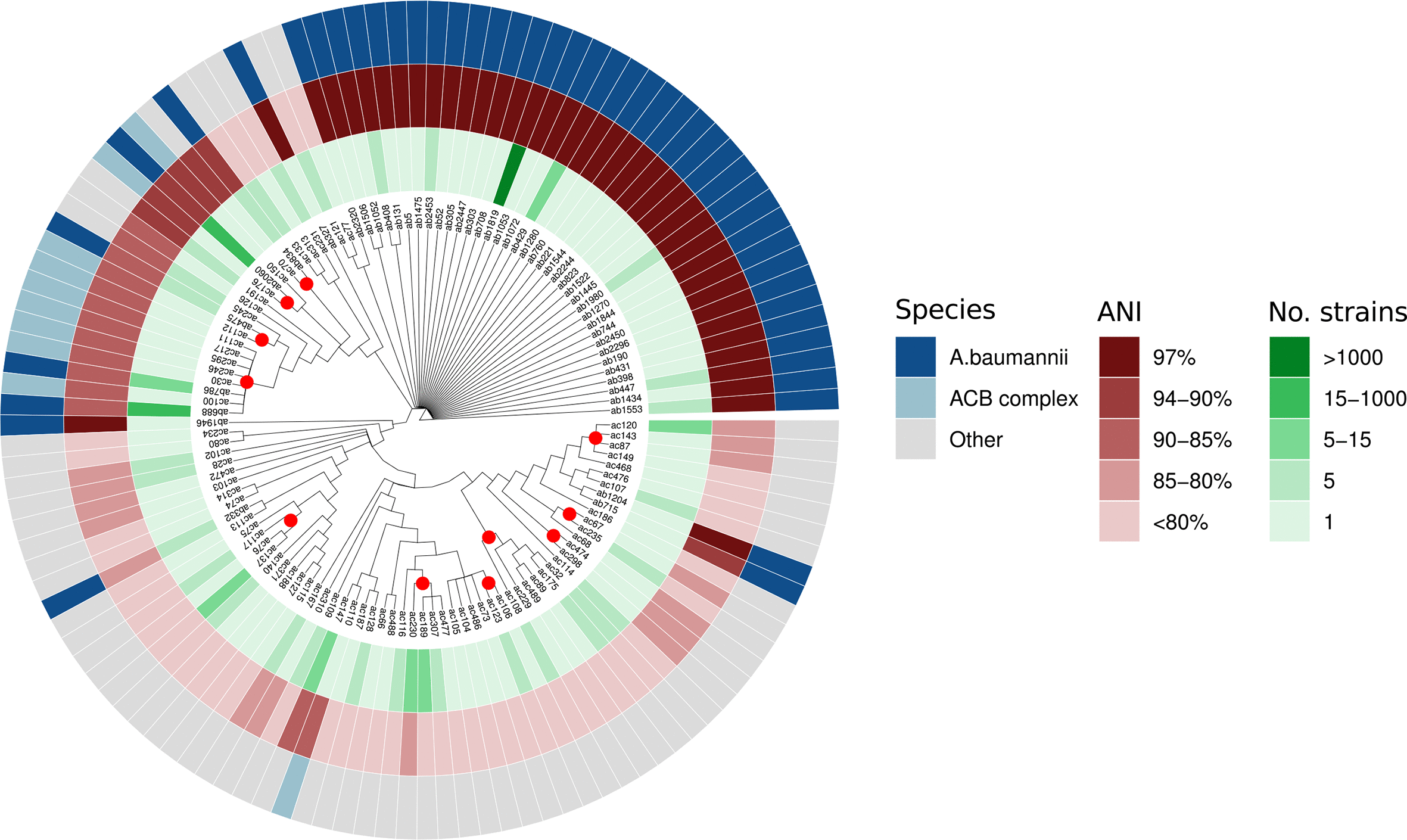
Filogenia molecular de bacterias del genero Acinetobacter analizadas en este trabajo. Cada nodo representa un grupo de genomas con el gen 16S ribosomal idéntico (en verde), y diferente similitud de secuencia genómica (en rojo). Imagen: Microbial Genomics.
La bacteria se puede diseminar a través del aire a distancias cortas y a través de la descamación de la piel de pacientes colonizados. Pero el modo de transmisión más común es a través de las superficies y material hospitalario y, sobre todo, a través de las manos del personal sanitario. La gran tasa de morbilidad y mortalidad que presenta A. baumannii se debe principalmente a que es una bacteria multirresistente, encontrándose cepas que resisten a la mayoría de antibióticos utilizados habitualmente. Además, hay que destacar que las infecciones de esta bacteria han aumentado desde que tuvo lugar la guerra de Iraq, y se cree que cepas infecciosas de esta bacteria provienen de heridas de soldados de esa contienda, lo que le ha llevado a que se la conozca desde entonces con el nombre de iraquibacteria.
Últimas publicaciones
Investigadores de la Universidad Pablo de Olavide colaboran en un estudio pionero liderado por el Centro de Investigación Príncipe Felipe que acelera la recuperación en animales tras una lesión medular y que tiene su base en la molécula AMPc.
Sigue leyendoEl estudio, publicado en la revista Bioorganic Chemistry, identifica moléculas capaces de bloquear una interacción clave en la progresión de este tipo de tumor. La investigación se ha centrado en el cáncer de mama triple negativo, un subtipo que no responde a los tratamientos hormonales convencionales ni a terapias dirigidas a otros receptores habituales, lo que limita gravemente las opciones terapéuticas.
Sigue leyendoInvestigadores del CSIC señalan que los cambios climáticos y el aporte de agua desde ríos y lagos explicarían los datos que muestran una cuenca marina casi vacía y, al mismo tiempo, llena de agua a finales del Mioceno.
Sigue leyendo



