Un nuevo modelo describe cómo las bacterias construyen el flagelo, el eficaz «propulsor» que les permite desplazarse
El grupo de investigación Genética del desarrollo de biofilms bacterianos, del Centro Andaluz de Biología del Desarrollo, ha establecido un modelo innovador que explica cómo las bacterias leen y ejecutan el manual para construir el flagelo, estructura esencial que permite que éstas se desplacen.
Fuente: Universidad Pablo de Olavide
Para sobrevivir, algunas bacterias se desplazan y colonizan nuevos lugares, ya sea en el medio ambiente o infectando a otros seres vivos, o bien escapan de ambientes hostiles, por ejemplo, debido a la presencia de un compuesto tóxico. El desplazamiento se convierte así en una condición esencial para la supervivencia de determinadas bacterias y, para ello, necesitan disponer de una estructura imprescindible, el flagelo, una perfecta y pequeña máquina molecular compuesta de varias partes que interactúan y contribuyen a la función básica, donde eliminar alguna de ellas interrumpiría las funciones de ese sistema.
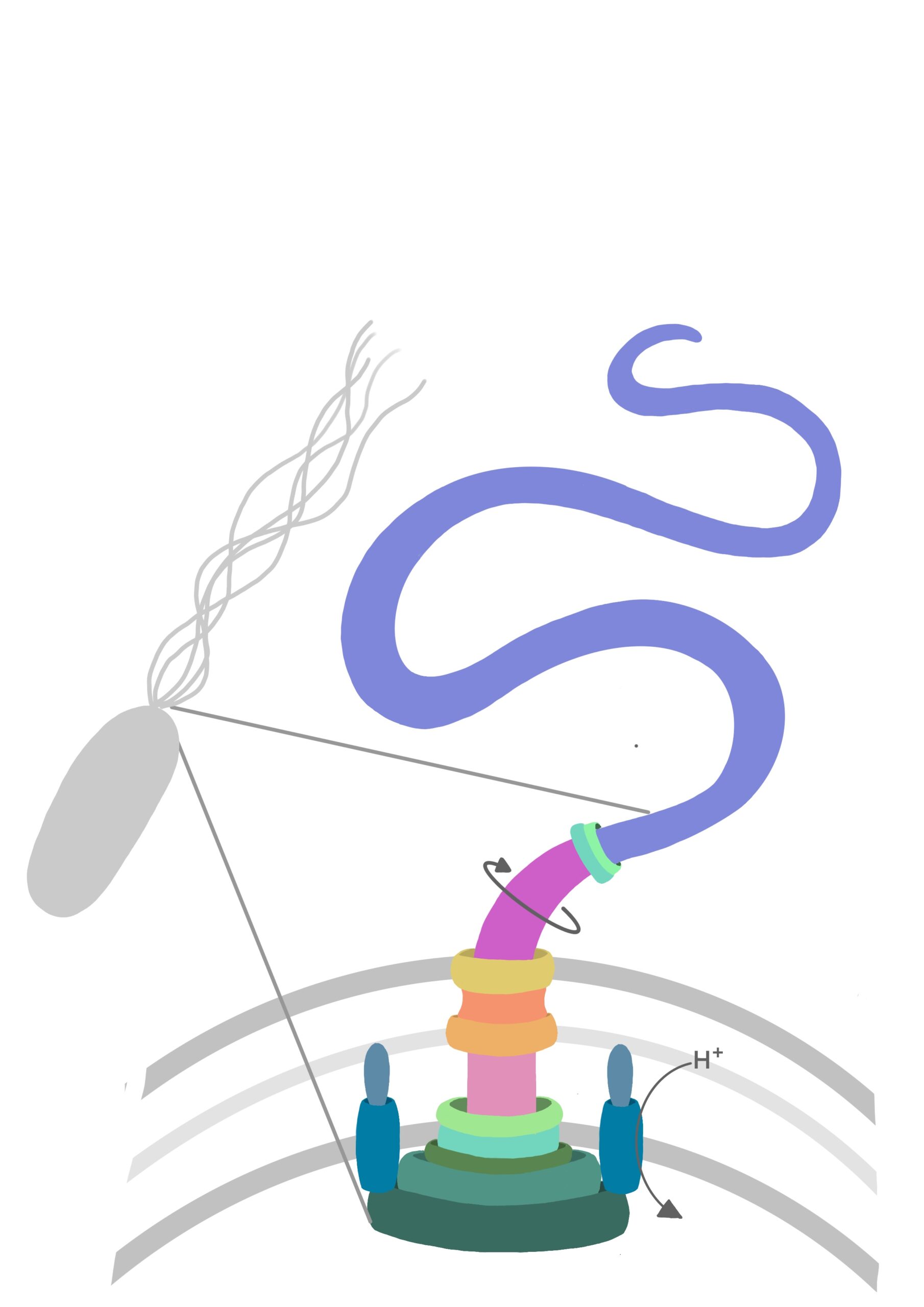
Estructura simplificada de un flagelo bacteriano. El motor está anclado a la pared celular de la bacteria, y es capaz de convertir la energía de una corriente eléctrica de protones en rotación. Contiene un interruptor para poder cambiar el sentido de giro. Esta rotación se transmite a través de un eje al filamento, que propulsa a la bacteria para poder desplazarse.
Estudiar cómo se comportan y funcionan las bacterias es determinante para conocer los numerosos procesos fundamentales para la vida en los que participan. Por ello, un grupo de investigación del Centro Andaluz de Biología del Desarrollo (CABD) ha establecido un modelo innovador que explica cómo las bacterias leen y ejecutan el manual para construir el flagelo, estructura esencial que permite que éstas se desplacen. El estudio, liderado por el investigador del Área de Microbiología de la Universidad Pablo de Olavide Fernando Govantes, ha sido publicado recientemente en la revista Environmental Microbiology.
Una máquina sofisticada
El flagelo bacteriano es la máquina más pequeña que existe, un dispositivo diminuto capaz de convertir una corriente eléctrica (de protones) en rotación, y permite propulsar a las bacterias a la mayor velocidad en relación a su tamaño registrada en un ser vivo: hasta 100 veces su longitud por segundo. Estructuralmente, es parecido a la hélice de un barco, está formado por un motor que proporciona energía para la rotación, un interruptor que cambia el sentido de giro, el eje de giro que conecta el motor con el filamento, y el propio filamento, un apéndice proteico que sobresale de la célula y que funciona como las palas de la hélice.
Está compuesto por más de 30 proteínas diferentes que se auto ensamblan de manera secuencial como piezas de la máquina. De igual manera, los genes que contienen las instrucciones en el ADN para fabricar estas proteínas, deben expresarse también en un orden determinado. A esta organización temporal en la ejecución de órdenes la llamamos ‘cascada flagelar’.
“Llevamos más de una década investigando la movilidad bacteriana utilizando como organismo modelo Pseudomonas putida, una bacteria de gran interés en biotecnología ambiental y agricultura ya que se asocia a las raíces de las plantas y promueve su crecimiento, a la vez que las protege de posibles patógenos”, afirma Fernando Govantes, investigador de la UPO y responsable del grupo ‘Genética del desarrollo de biofilms bacterianos’ del CABD, centro mixto del Consejo Superior de Investigaciones Científicas (CSIC), la Universidad Pablo de Olavide y la Junta de Andalucía.
P. putida tiene un penacho de entre 5-7 flagelos en un único polo de la célula que utiliza para desplazarse. En esta bacteria, las instrucciones para fabricar las piezas del flagelo se encuentran agrupadas juntas en el genoma, como si fueran un manual.
La superación de un modelo simplista
En este trabajo, el equipo ha combinado por primera vez datos de secuenciación, análisis computacionales de la conservación y organización de los genes flagelares entre distintas especies de Pseudomonas y trabajo experimental, para explicar de manera realista en qué orden se ejecutan todas estas instrucciones para construir los flagelos bacterianos. El modelo simplista que la comunidad científica asumía hasta ahora se basaba en que los genes flagelares se expresan por grupos muy definidos en 3-4 oleadas de manera secuencial: serían los saltos de la cascada. Primero se expresan las proteínas que dan la orden a la célula para empezar a construir el flagelo. Estas proteínas se encargan de hacer que se exprese el núcleo central de la máquina que se inserta dentro de la pared celular. Más tarde, otras dan la orden para construir el filamento que se extiende hacia afuera de la bacteria.
“Sin embargo, con la estrategia que hemos seguido integrando resultados de análisis diferentes, hemos descubierto en P. putida que la realidad es mucho más compleja: no solo existen estos saltos en la cascada, sino que nos encontramos con un segundo nivel de regulación superpuesto: proteínas que ordenan la construcción de las últimas piezas de la máquina también ordenan que se fabriquen más piezas de las iniciales. Además, cuando se escriben las instrucciones para hacer el filamento, se escriben a la vez las que hacen que la célula vuelva a iniciar la síntesis del flagelo desde el principio”, explica Fernando Govantes.
En contraposición, el equipo también ha encontrado otras proteínas que se expresan a la vez que las del núcleo del flagelo, y que impiden la fabricación de más piezas iniciales. Estos bucles facilitan la interrupción de la fabricación de componentes flagelares ya ensamblados, y también la reiniciación de la síntesis de nuevos flagelos para las células hijas cuando se acerca la división celular.
“La gran conservación entre diferentes especies bacterianas sobre cómo se organizan estos bloques de instrucciones en el genoma y las secuencias de ADN que reconocen las proteínas que las ejecutan, nos indica que esta nueva forma de concebir la cascada flagelar se aplica, como mínimo, al resto de especies de Pseudomonas que habitan en suelos y sobre las superficies de las plantas”, concluye Fernando Govantes.
Referencia bibliográfica:
Antonio Leal-Morales, Marta Pulido-Sánchez, Aroa López-Sánchez, Fernando Govantes (2021) Transcriptional organization and regulation of the Pseudomonas putida flagellar system. Environmental Microbiology. https://doi.org/10.1111/1462-2920.15857
Publicaciones relacionadas
Un equipo de investigación de la Estación Experimental del Zaidín del CSIC en Granada ha descubierto una proteína microbiana que reconoce varios compuestos de origen vegetal para orientar su desplazamiento hacia el lugar donde encontrar nutrientes. El hallazgo se enfoca hacia al diseño de nuevas estrategias biotecnológicas para favorecer la colonización de plantas por bacterias que promueven su crecimiento y las protegen frente a enfermedades.
Sigue leyendoUn equipo de investigación de la Universidad de Jaén ha confirmado en ensayos con ratones que una dieta basada en AOVE permite una mejor regulación de la microbiota del intestino que otras grasas refinadas o mantequilla. Además, ha verificado que la función de estos microorganismos se altera según el tipo de aceite que se consume y se relaciona directamente con el síndrome metabólico, un grupo de factores de riesgo para enfermedades cardiacas, diabetes y otros problemas de salud.
Sigue leyendoUn equipo de investigación de la Estación Experimental del Zaidín (CSIC) de Granada ha participado en la creación de la herramienta gratuita PADLOC. Ésta sirve como un ‘catálogo’ de consulta, donde los investigadores introducen una secuencia del genoma de la bacteria y el programa les indica qué sistema de protección posee el microorganismo.
Sigue leyendo




