Describen por qué las enzimas de Salmonella atacan con distinta virulencia según los casos
Investigadores de la Universidad de Sevilla, en colaboración con las universidades de Zaragoza y Kansas (EEUU), han conseguido describir procesos hasta ahora desconocidos que permiten comprender la virulencia de una infección por Salmonella. En concreto, su estudio ha demostrado que, en determinados factores de virulencia de Salmonella, un simple aminoácido es responsable de determinar qué proteínas de la célula infectada son modificadas.
Fuente: Universidad de Sevilla
Investigadores de la Universidad de Sevilla, en colaboración con las universidades de Zaragoza y Kansas (EEUU), han conseguido describir procesos hasta ahora desconocidos que permiten comprender la virulencia de una infección por Salmonella. En concreto, su estudio, publicado recientemente en la revista Chemical Science, ha demostrado que, en determinados factores de virulencia de Salmonella, un simple aminoácido es responsable de determinar qué proteínas de la célula infectada son modificadas.
Escherichia coli y Salmonella entérica son enterobacterias responsables de multitud de casos de infecciones de origen alimentario, típicamente asociadas a diarrea, fiebre, y nauseas, con distintos grados de severidad.
Escherichia coli y Salmonella entérica son enterobacterias responsables de multitud de casos de infecciones de origen alimentario, típicamente asociadas a diarrea, fiebre, y nauseas, con distintos grados de severidad. Estos microbios han desarrollado a lo largo de su evolución todo un arsenal estratégico que emplean durante el proceso de infección para poder resistir los mecanismos naturales de defensa de nuestras células, así como para favorecer su diseminación en los tejidos infectados (invasión microbiana). Una parte importante de dicho arsenal de ataque lo constituyen los denominados ‘factores de virulencia’, que son moléculas, típicamente proteínas, que utilizan dichos microbios para debilitar la respuesta inmune natural de las células infectadas hacia el invasor, favoreciendo así su diseminación en el organismo infectado.
Entre el conjunto de factores de virulencia se destacan las propias enzimas bacterianas, que inducen modificaciones químicas en las células infectadas, facilitando la infección y posterior invasión microbiana. Un aspecto importante es que, para que se produzcan dichas modificaciones en la célula, es necesario que dichas enzimas se unan a determinadas proteínas de la célula infectada, de modo que una forma muy prometedora de evitar la invasión microbiana es la inhibición de dicha interacción, es decir, evitar que dichas moléculas, del invasor y de la célula infectada, se unan. Esto se puede realizar mediante el uso de moléculas diseñadas con el fin de ocupar el sitio de la enzima bacteriana al que se une la proteína celular. Sin embargo, para que el diseño de estas moléculas (denominadas inhibidores) sea eficaz, es fundamental conocer con detalle las características tridimensionales de dichas enzimas, así como cuáles de los componentes fundamentales de estas enzimas, es decir, los aminoácidos, son los responsables de la actividad enzimática que les confiere virulencia.
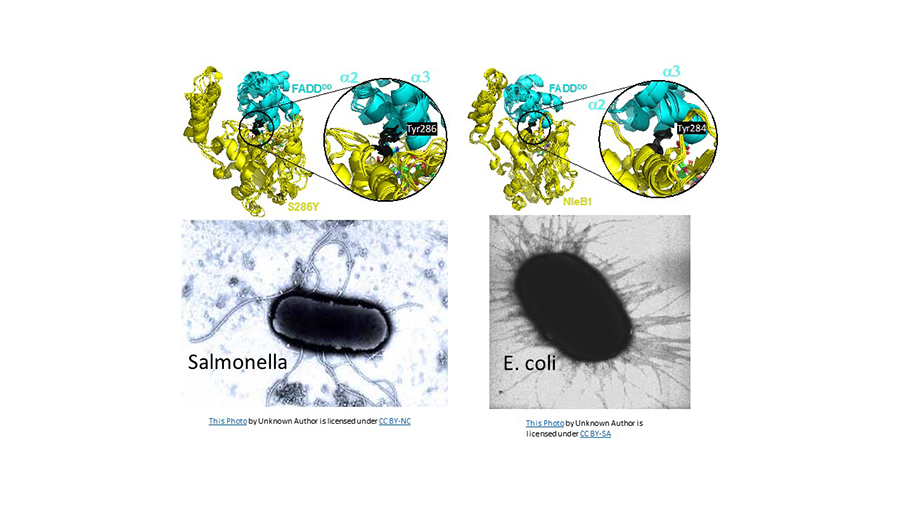
“Nuestro interés se centraba en analizar qué aminoácidos de ciertos factores de virulencia de Salmonella eran responsables de que estas enzimas fuesen más selectivas, es decir, que modificasen un menor número de proteínas de la célula infectada en comparación con enzimas homólogas de Escherichia coli. Nos enfocamos en enzimas denominadas glicosiltransferasas, que son un tema central en nuestro laboratorio, y observamos, mediante estudios de actividad con distintos mutantes que, tan solo cambiando un aminoácido, es decir, una mutación simple, dichas enzimas de Salmonella recuperaban la capacidad de afectar a un mayor número de sustratos y mostrar así una actividad parecida a las enzimas homólogas de Escherichia coli”, explica el profesor de la Universidad de Zaragoza Ramón Hurtado-Guerrero.
El estudio es de elevado interés, puesto que no solamente abre las puertas a entender las bases moleculares de por qué las enzimas de Salmonella tienen ciertos sustratos humanos específicos y no son de amplio espectro como la enzima de E. coli. Además, el trabajo ha permitido determinar cómo la patogenicidad de Salmonella entérica se puede variar solo con simples cambios en estas enzimas, haciendo que sea menos o más patógena según la mutación.
“Un aspecto importante era dilucidar cuál es el impacto que tiene la mutación simple en la unión entre el factor de virulencia y la proteína de la célula infectada”, explica el investigador de la Universidad de Sevilla Jesús Angulo. “En nuestro caso, estudiamos por qué la mutación simple de la enzima denominada SseK1 de Salmonella hace que ésta sea activa para modificar una proteína determinada, denominada FADD, que se encuentra en la célula infectada, que en condiciones “normales” no modificaría. Para ello, llevamos a cabo cálculos de dinámica molecular que demostraron que dicha mutación, que no está en el “sitio activo” de la enzima, hace que la enzima y la proteína sustrato se unan más fuertemente, ya que el aminoácido mutado mejora el ajuste entre las superficies de contacto de ambas moléculas (algo así como si se mejora el contorno de una llave para que se ajuste mejor a la cerradura). Además, dichas simulaciones de dinámica molecular demostraban que la mutación simple afectaba favorablemente a la dinámica del sitio catalítico, de forma que se favorece la modificación química de la proteína hospedadora de la célula infectada. Es un ejemplo muy llamativo de cómo una simple mutación puede ampliar el espectro de acción de una enzima, mediante una mejora simultánea de la afinidad de la unión y de la dinámica de los residuos implicados en el proceso catalítico”, explica Jesús Angulo.
Estos resultados son de elevado interés en la compresión molecular de la acción de los factores de virulencia bacterianos y pueden inspirar el desarrollo de inhibidores como terapias antibacterianas alternativas a los antibióticos.
Referencia bibliográfica:
NleB/SseK-catalyzed arginine-glycosylation and enteropathogen virulence are finely tuned by a single variable position contiguous to the catalytic machinery; Ana García-García, Thomas Hicks, Samir El Qaidi, Congrui Zhu, Philip R. Hardwidge, Jesús Angulo, Ramon Hurtado-Guerrero; Chemical Science, agosto 2021
Últimas publicaciones
Investigadores de la Universidad Pablo de Olavide colaboran en un estudio pionero liderado por el Centro de Investigación Príncipe Felipe que acelera la recuperación en animales tras una lesión medular y que tiene su base en la molécula AMPc.
Sigue leyendoEl estudio, publicado en la revista Bioorganic Chemistry, identifica moléculas capaces de bloquear una interacción clave en la progresión de este tipo de tumor. La investigación se ha centrado en el cáncer de mama triple negativo, un subtipo que no responde a los tratamientos hormonales convencionales ni a terapias dirigidas a otros receptores habituales, lo que limita gravemente las opciones terapéuticas.
Sigue leyendoInvestigadores del CSIC señalan que los cambios climáticos y el aporte de agua desde ríos y lagos explicarían los datos que muestran una cuenca marina casi vacía y, al mismo tiempo, llena de agua a finales del Mioceno.
Sigue leyendo



