Descubren que genes clave para frenar el desarrollo de linfomas adquieren mutaciones en los sitios que regulan las “señales de corte y empalme”
El equipo de investigación del profesor de la Universidad de Granada Pedro Medina Vico ha analizado datos genéticos de más de mil quinientos pacientes de linfoma difuso de células B grandes, en busca de nuevas mutaciones que puedan estar relacionadas con la enfermedad.
Fuente: Universidad de Granada
Una investigación de la Universidad de Granada ha identificado nuevas mutaciones relacionadas con el linfoma que ayudan a entender mejor cómo se producen estos tumores. El estudio, en el que ha participado el Instituto de Investigación Biosanitaria de Granada (ibs.GRANADA) y el Centro Pfizer-Universidad de Granada-Junta de Andalucía de Genómica e Investigación Oncológica (Genyo), lo ha realizado el equipo del profesor de la UGR Pedro Medina Vico, del Departamento de Bioquímica y Biología Molecular I, y ha tenido como primeros autores a los investigadores Álvaro Andrades Delgado y Juan Carlos Álvarez Pérez.
El linfoma es un tipo de cáncer que afecta a células del sistema inmunitario denominadas linfocitos. A diferencia de las leucemias, que también se producen mediante tumores en las células sanguíneas, los linfomas generan tumores sólidos que se manifiestan en el sistema linfático. Según la Sociedad Española de Oncología Médica, en 2021 se diagnosticaron más de diez mil nuevos casos de linfoma en España, siendo el linfoma difuso de células B grandes (LDCBG) el subtipo más frecuente y uno de los de peor pronóstico. El LDCBG puede afectar a las personas de cualquier edad, pero generalmente ocurre en las más mayores. El LDCBG es uno de los linfomas más agresivos y de peor pronóstico. Alrededor del 35-45% de los pacientes no sobreviven cinco años después de su diagnóstico.
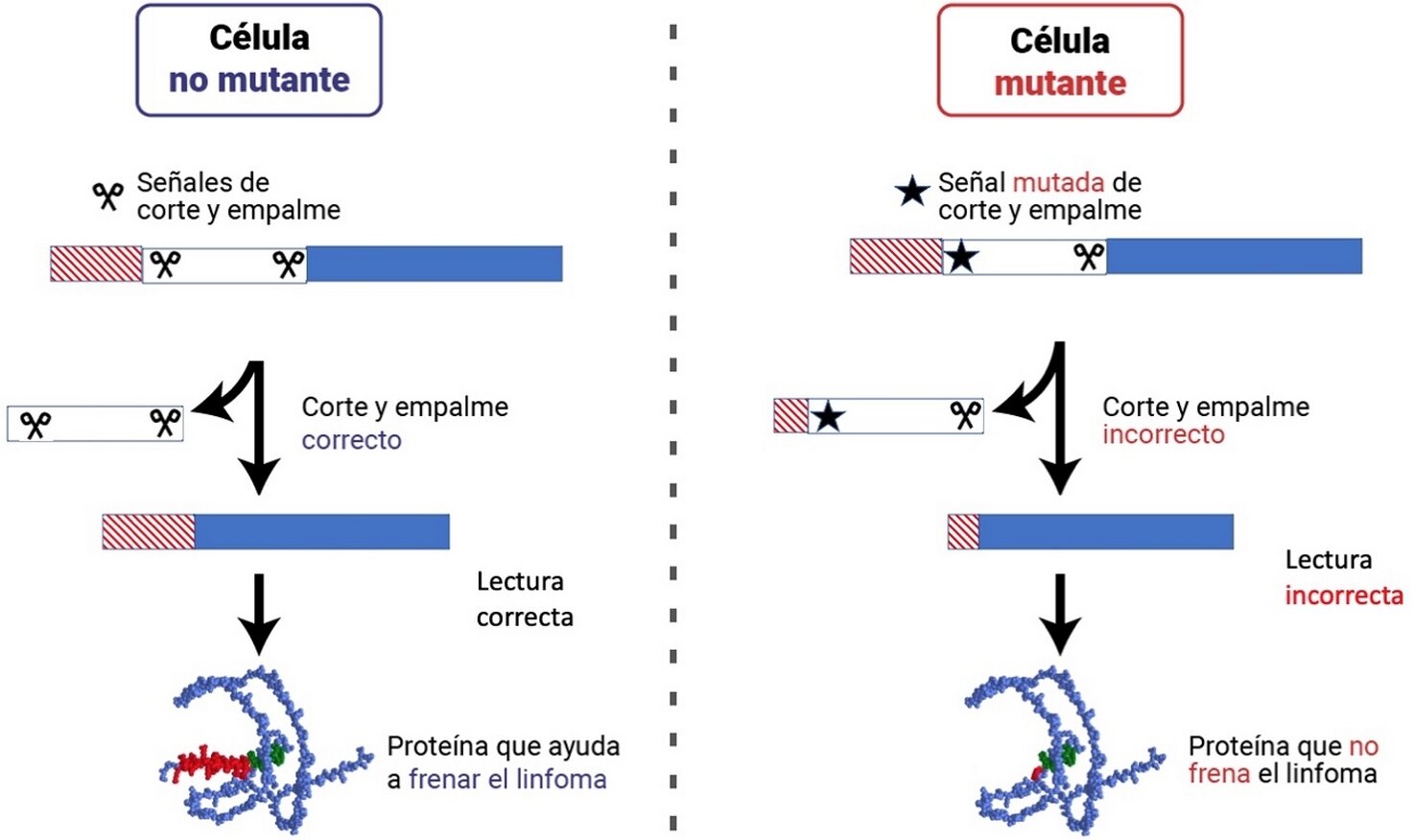
Los investigadores del grupo del profesor Medina han analizado datos genéticos de más de mil quinientos pacientes de LDCBG, en busca de nuevas mutaciones que puedan estar relacionadas con la enfermedad. Gracias a ello, han conseguido observar que, hasta ahora, habían pasado desapercibidas las acumulaciones de mutaciones en regiones específicas de ADN llamadas “señales de corte y de empalme” y que estaban presentes en genes importantes que frenan el desarrollo del linfoma.
“Los genes humanos tienen almacenada la información de una manera un tanto particular. Esto se debe a que codifican la información de forma fragmentada, ya que existen regiones que no se deben leer y han de cortarse y eliminarse para que se pueda generar un producto adecuado. Si no se realizan estos cortes y empalmes de información de manera precisa no se crean productos funcionales” comenta el profesor Medina.
“Así, en este trabajo hemos descubierto que se acumulan mutaciones que impiden que se genere un corte y empalme correcto cuando se leen genes importantes para frenar el desarrollo de linfomas”, añade Pedro Medina. El hallazgo tiene relevancia desde un punto de vista clínico, porque algunos de los genes identificados por los autores (como CD79B) están cobrando especial importancia en los últimos años, ya que sus mutaciones se han propuesto para el diagnóstico molecular de linfomas. Además, estas mutaciones pueden estar relacionadas con la respuesta del paciente a la terapia estándar, y se está estudiando su relación con la respuesta a nuevos tratamientos experimentales de última generación. Por ello, es especialmente importante identificar todas las alteraciones moleculares relacionadas con el LDCBG para, en un futuro, contar con métodos más precisos y personalizados para diagnosticar y elegir el tratamiento más eficaz para cada paciente.
Referencia bibliográfica:
Andrades Delgado, Alvaro, Juan Carlos Álvarez-Pérez, Juan Rodrigo Patiño-Mercau, Marta Cuadros, Carlos Baliñas-Gavira, y Pedro P Medina. «Recurrent splice site mutations affect key diffuse large B-cell lymphoma genes». Blood, 5 de enero de 2022, blood.2021011708. https://doi.org/10.1182/blood.2021011708
Últimas publicaciones
El atlas define con precisión todos los tipos de células germinales masculinas que participan en la espermatogénesis de esta especie y caracteriza los alrededor de 160 genes clave que dirigen cada etapa del proceso.
Sigue leyendoUn equipo de investigación de la Universidad de Huelva ha extraído el metal contenido en los lodos procedentes de la depuración de agua industrial para devolverlo al circuito productivo con la misma utilidad y eficacia que el producto original. La técnica permite aprovechar la mayor parte del material, que antes se desechaba y disminuir el volumen enviado a vertedero.
Un equipo de investigación de la Universidad de Cádiz ha desarrollado un modelo matemático para estimar con mayor exactitud la cantidad de sedimento necesario para regenerar las costas. La propuesta permite ajustar mejor el volumen añadido al litoral, reduciendo costes y respetando la composición natural del ecosistema.
Sigue leyendo