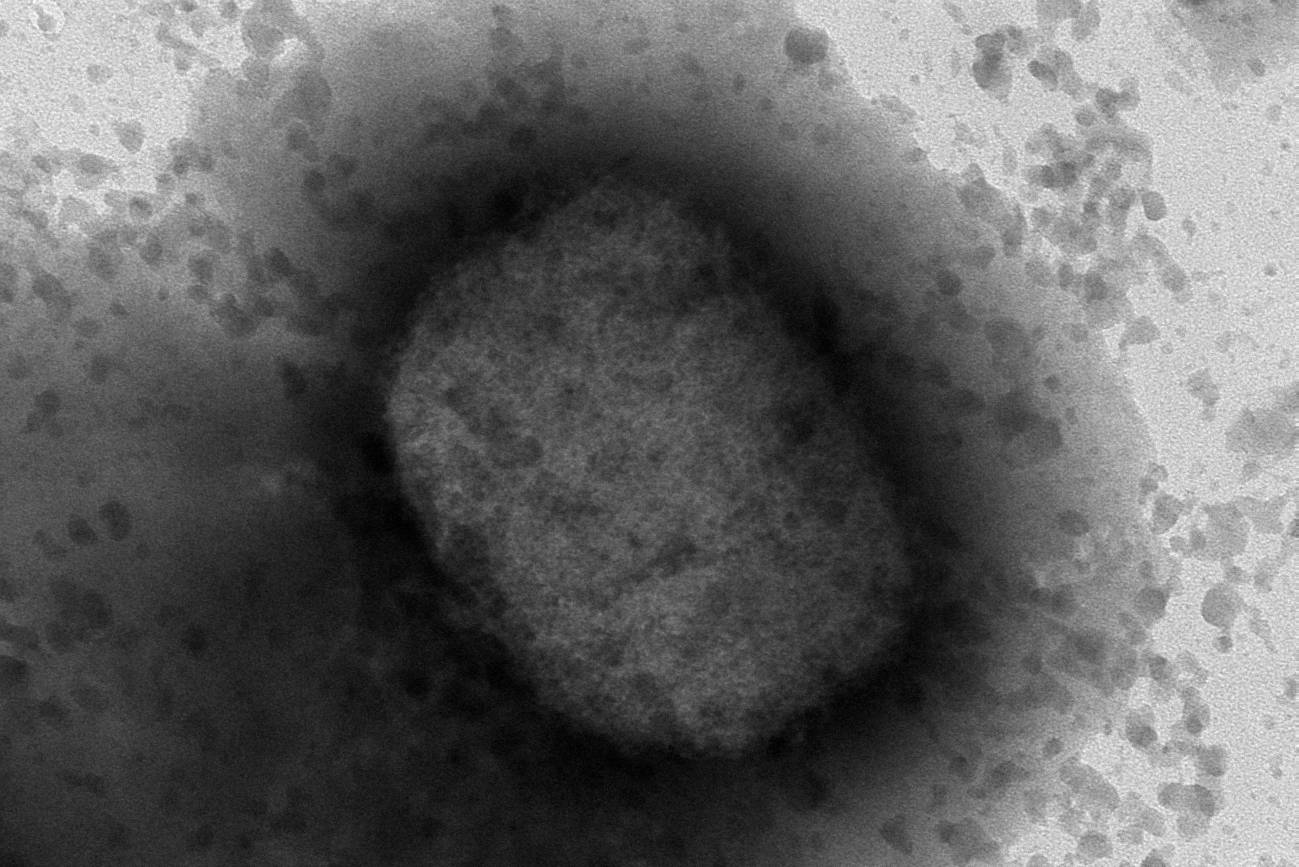
Obtienen la secuencia del 100 % del genoma del virus de la viruela del mono
Un equipo del Laboratorio de Arbovirus y de las Unidades de Genómica y Bioinformática del Instituto de Salud Carlos III ha logrado la secuenciación completa del virus de la viruela del mono, un logro que permitirá hacer análisis filogenéticos más avanzados para obtener datos sobre su comportamiento y comprender mejor su origen, circulación y difusión.
Fuente: Agencia SINC
Investigadores del Instituto de Salud Carlos III (ISCIII) han logrado el primer borrador de secuencia completa del virus de la viruela del mono, obtenido del análisis genómico de muestras de 23 pacientes. Esta labor de secuenciación masiva permite confirmar que es la variedad de África Occidental la causante de este brote.
Adicionalmente, la secuenciación ha alcanzado una cobertura del 100 % de los 190.000 pares de bases del genoma de este virus, lo cual abre la posibilidad de estudios filogenéticos más avanzados que permitirán obtener información adicional sobre su comportamiento y comprender mejor su origen, circulación y difusión. Se trata de una de las secuencias más completas obtenidas hasta el momento.
La secuenciación llevada a cabo a través del Laboratorio de Arbovirus del ISCIII, en conjunto con las unidades de Genómica y Bioinformática del, ha contado con las referencias publicadas en los últimos días por otros países (Bélgica, Alemania, Portugal y EEUU), y se ha basado en una tecnología genómica de nueva generación mapeada contra genoma de referencia, a lo que se ha sumado un análisis complementario de las muestras mediante una técnica conocida como ensamblado de novo. Las secuencias en crudo se terminaron de obtener el pasado lunes por la noche y el análisis de computación se ha realizado en las últimas 36 horas.
Los resultados señalan que las muestras secuenciadas parecen pertenecer al mismo brote detectado en otros países europeos, ya que los genomas obtenidos apenas difieren de los ya secuenciados en otros países; en concreto, el análisis realizado en el ISCIII concluye que hay menos de 5 SNP -secuencias genéticas denominadas cambios de nucleótidos sencillos- de diferencia respecto a la secuenciación llevada a cabo en Alemania.
La secuenciación confirma que el virus de la viruela de los monos del brote que se está produciendo en España es del clado de África Occidental, que es el de menor virulencia entre los conocidos y el que se ha identificado por el momento en la mayoría de los países fuera de África implicados en este brote. Los clados son grupos filogenéticos que definen la evolución biológica de un organismo, que explican cómo actúa y se comporta, y en ellos se pueden observan las diferencias genéticas de los virus circulantes.
Tras obtener la secuencia completa del virus, varios equipos de investigadoras del ISCIII están llevando a cabo análisis filogenéticos para conocer la relación entre las muestras españolas y las de otros países.
La información obtenida se enfrentará a las ya conocidas y depositadas en bases de datos internacionales para evaluar el grado de identidad y, en su caso, la localización de las diferencias que pudieran existir entre la secuencia española y los demás datos internacionales. Todo ello permitirá realizar los estudios de trazabilidad del brote, así como identificar potencialmente su origen.
Últimas publicaciones
Para conmemorar el Día de las Matemáticas (14 de marzo), la Fundación Descubre organiza estos encuentros donde investigadores […]
Sigue leyendoLa participación está abierta a: municipios de menos de 50.000 habitantes, así como a colectivos sociales y en riesgo de exclusión.
Sigue leyendoUn equipo de la Universidad de Sevilla descubre una diversidad genética inesperada en un grupo de tomillos ibéricos. El trabajo, basado en técnicas genómicas de última generación, revela linajes ocultos y plantea nuevos retos para la conservación de estas plantas tan emblemáticas.