Revelan que los aminoácidos actúan como moléculas señal en todo el Árbol de la Vida
Un estudio en el que participa el grupo de Microbiología Ambiental y Biodegradación de la Estación Experimental del Zaidín (EEZ/CSIC) corrobora que los aminoácidos, además de componentes básicos de la vida, se reconocen como señales en bacterias, arqueas y organismos eucariotas, incluyendo células humanas, a través de un dominio proteico sensor que está conservado en todos los linajes de la vida. Este trabajo puede abrir nuevas vías para explorar el papel de receptores de aminoácidos en el desarrollo de procesos virulentos.
Fuente: CSIC Andalucía
La Estación Experimental del Zaidín (EEZ-CSIC) colabora en una investigación internacional multidisciplinar con “The Ohio State University” (Estados Unidos) y “University College London” (Reino Unido), que ha permitido identificar un dominio proteico que actúa como un sensor universal capaz de reconocer aminoácidos. Este dominio, denominado dCache_1AA, se encuentra en proteínas presentes en todos los linajes de la vida, desde bacterias a humanos. La investigación deriva del análisis detallado de las estructuras tridimensionales de distintas proteínas receptoras bacterianas definidas en colaboración con el Laboratorio de Estudios Cristalográficos del Instituto Andaluz de Ciencias de la Tierra (IACT-CSIC-UGR).

Tino Krell, Elizabet Monteagudo-Cascales y Miguel A. Matilla / Foto: Unidad de Cultura Científica EEZ-CSIC.
Todos los seres vivos tienen proteínas sensoras que detectan una gran diversidad de cambios ambientales, para adaptar su metabolismo y fisiología a dichas alteraciones. Sin embargo, se desconoce la función concreta de la mayoría de estos receptores. Mediante la combinación de análisis bioinformáticos, bioquímicos y de biología estructural, se han identificado miles de receptores que reconocen aminoácidos a través del dominio dCache_IAA. Los aminoácidos son componentes básicos de la vida: son los “bloques” que conforman las proteínas y están involucrados en una gran variedad de procesos celulares. Este estudio, publicado en la prestigiosa revista Proceedings of the National Academy of Science (PNAS), confirma que además los aminoácidos pueden funcionar como señales en bacterias, arqueas y organismos eucariotas, incluyendo células humanas, a través de un mecanismo universal de reconocimiento.
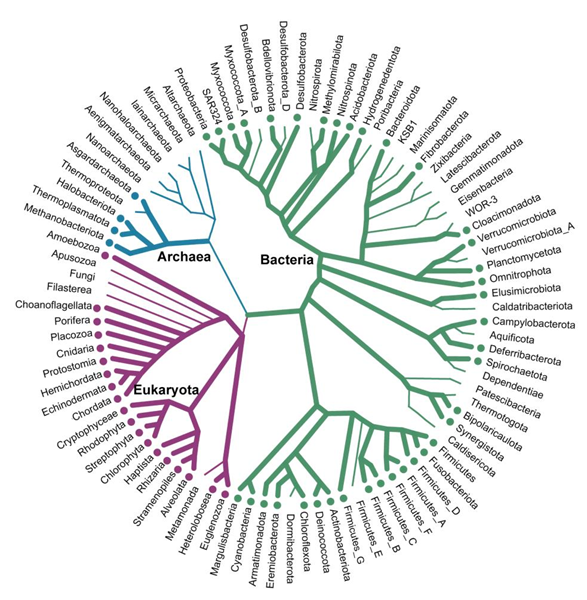
Pie de figura: Identificación de un dominio de unión a aminoácidos a lo largo del Árbol de la Vida. Los puntos indican la presencia del dominio de reconocimiento de aminoácidos en distintos grupos taxonómicos / Krell Laboratory EEZ-CSIC.
El grupo de Microbiología Ambiental y Biodegradación de la EEZ-CSIC de Granada ha confirmado en esta publicación la presencia de estos receptores en diversas bacterias patógenas, como Yersinia pestis (agente causal de la peste) y Vibrio cholerae (agente causal del cólera). Tino Krell, Profesor de Investigación en la Estación Experimental del Zaidín afirma que “este trabajo puede abrir nuevas vías para explorar la posible implicación de estos receptores en el desarrollo de procesos virulentos. En humanos, se ha detectado el dominio dCache_IAA en proteínas que son diana de medicamentos dirigidos al tratamiento del dolor y de trastornos neurobiológicos”
Dado que la gran mayoría de las proteínas sensoras y las señales que reconocen permanecen sin ser estudiadas, esta investigación ofrece nuevos enfoques para identificar los mecanismos de señalización en organismos a lo largo del Árbol de la Vida.
Referencia bibliográfica:
Vadim M. Gumerov, Ekaterina P. Andrianova, Miguel A. Matilla, Karen M. Page, Elizabet Monteagudo-Cascales, Annette C. Dolphin, Tino Krell & Igor B. Zhulin. (2022). Amino acid sensor conserverd from bacteria to humans. PNAS.
Contacto:
Tino Krell – tino.krell@eez.csic.es – Tel: 958526579
Miguel A. Matilla – miguel.matilla@eez.csic.es – Tel: 958526506
Elizabet Monteagudo-Cascales – elizabet.monteagudo@eez.csic.es – Tel: 958526452
Últimas publicaciones
Para conmemorar el Día de las Matemáticas (14 de marzo), la Fundación Descubre organiza estos encuentros donde investigadores […]
Sigue leyendoLa participación está abierta a: municipios de menos de 50.000 habitantes, así como a colectivos sociales y en riesgo de exclusión.
Sigue leyendoUn equipo de la Universidad de Sevilla descubre una diversidad genética inesperada en un grupo de tomillos ibéricos. El trabajo, basado en técnicas genómicas de última generación, revela linajes ocultos y plantea nuevos retos para la conservación de estas plantas tan emblemáticas.




